Einführung
Wer sind wir? Woher kommen wir? Das sind Fragen, die jeder irgendwann stellt. Wir alle lernen etwas über die Geschichte unseres Landes in der Schule. Aber die Geschichte hat ihre Grenzen. Sie wird uns nicht sagen, was jeden von uns unterscheidet.
Mit Hilfe der Wissenschaft können wir jetzt ursprünglichen Herkünfte der patrilinearen Vorfahren bestimmen, indem wir die DNS des väterlich vererbten Y-Chromosoms (kurz: Y-DNS) testen.
Geschichtsbücher untersuchen selten persönliche Genealogien, außer für königliche Familien. Aber auch königliche Familien sind nicht immun gegen Nicht-Vaterschaftsereignisse. Zum Beispiel ergaben Y-DNS-Tests, dass Napoleon III nicht die gleiche väterliche Abstammung (I2a2a) wie sein väterlicher Oheim Napoleon I (E1b1b) hat. Er wurde anscheinend aus einer unehelichen Verbindung heraus geboren. Aber der Hauptzweck der historischen Populationsgenetik besteht darin, die entfernten Vorfahren zurückzuverfolgen, Hunderte, Tausende oder sogar Zehntausende von Jahren zurückzugehen. Die erstaunliche Sache mit der modernen Genetik ist, dass es möglich ist, zu wissen, woher unsere Vorfahren in verschiedenen Perioden der Geschichte kamen, zumindest hinsichtlich der rein väterlichen Linie. Stammten wir ab von Kelten, Slawen, Römern, germanischen Stämmen, Juden, Phöniziern, alten Griechen? Bis vor wenigen Jahrzehnten wäre es eine Science-Fiction gewesen, zu wissen, wo die Vorfahren vor 2000 oder 3000 Jahren lebten. Das ist es nicht mehr.
Wie können wir unsere Herkunft mit DNS verfolgen?
Wir haben 23 Chromosomenpaare. Das letzte Paar ist X-Y für Männer und X-X für Frauen. Das Y-Chromosom ist der einzige Teil der DNS, der während der Fortpflanzung nicht rekombiniert wird. Dies liegt daran, dass die X- und Y-Chromosomen unterschiedlich lang sind und nicht miteinander verschmelzen können. Dies erklärt, warum das Y-Chromosom (das wir kurz "Y-DNS" nennen werden) von Generation zu Generation praktisch unverändert bleibt und daher bei allen Menschen identisch ist, die von einem nicht allzu weit entfernten gemeinsamen Vorfahren abstammen (einige tausend Jahre).
Das Y-Chromosom ist eine Sequenz von 59 Millionen Zeichen. Einige Kopierfehler (Mutationen) passieren in jeder Generation, wie auf allen anderen Chromosomen. Jede Mutation, die in jedem neuen Individuum auftritt, wird von seinen Nachkommen geerbt. Durch Auflisten dieser Mutationen und durch Aufsummieren aller in einem Individuum gefundenen Mutationen ist es möglich, seine Genealogie zu verfolgen und die Anzahl der Generationen zu bestimmen, die es von irgendeinem anderen Mann in der Welt trennt.
Jeder Mensch wird daher die gleiche Y-DNS haben, wie sein Vater, Brüder, Söhne, väterlicher Großvater, und so weiter, ein paar Mutationen. Dies ist der Grund, warum alle Männer, die von einem gleichen patrilinearen Vorfahren abstammen (und daher denselben Nachnamen tragen), die gleiche Reihe von Mutationen teilen, die von all ihren gesammelten väterlichen Vorfahren seit Tausenden von Jahren ererbt wurden. Alle Männer mit dem gleichen Satz an Mutationen, die von vor x-tausenden von Jahren ererbt wurden, können daher als die selbe Familie klassifiziert werden, die Populationsgenetiker eine Haplogruppe nennen. Die Menschheit ist so in einem einzigen großen Stammbaum durch das Y-Chromosom vereint, mit vielen Verzweigungen (Haplogruppen) und Verzweigungen (Unterkladen), die sich über die Jahrtausende entwickelt haben. Der letzte gemeinsame menschliche paternale Vorfahre der gesamten Menschheit (auch bekannt als Y-chromosomaler Adam) lebte in Afrika vor mindestens 300.000 Jahren.
Diese Mutationen, die in jeder Generation auftreten, sind als Einzelnukleotid-Polymorphismus (abgekürzt SNP auf Englisch für Single Nucleotide Polymorphism) bekannt. Sie sind in der Reihenfolge nach dem Zeitpunkt ihrer Entdeckung numeriert.
| Mehr über DNA und SNPs |
|---|
|
Unsere DNS liest sich wie ein Buch in einem Alphabet mit vier Buchstaben: A, C, G und T. Sie treten immer paarweise auf, A mit T und G mit C. Solche Paare heißen "Basenpaare", auch bekannt als Nukleobasen. Ein SNP ist eine Mutation in einem solchen Basenpaar, zum Beispiel ein C ist ersetzt durch ein A. Unser Genom ist in 46 Chromosomen unterteilt, die als Bände einer Enzyklopädie angesehen werden können. Jedes Chromosom enthält Hunderte oder Tausende von Genen, die die Kapitel bilden. Insgesamt gibt es 3.000 Millionen Basenpaare. Wenn eine Mutation auftritt, kann sie den Auswirkung eines Gens verändern, aber nicht immer, da einige Mutationen still oder synonym sind. Das Y-Chromosom besteht aus 59 Millionen Nukleobasen. Ab Anfang 2017 wurden 55.000 SNPs identifiziert, um die verschiedenen väterlichen Abstammungslinien in der Welt zu unterscheiden.
Wenn sie entdeckt werden, erhalten SNPs eine Referenznummer, die mit Rs beginnt und deren Nummer in der Regel in die Millionen geht. SNP werden für das gesamte Genom verwendet, nicht nur für das Y-Chromosom. Um das genetische Risiko für die Entwicklung einer Krankheit zu beurteilen, werden Ärzte SNP betrachten, von denen bekannt ist, dass sie mit einer bestimmten Krankheit assoziiert sind, obwohl sich kaum eine auf dem Y-Chromosom befindet. Online-Datenbanken wie SNPedia erlauben Menschen, die ihr Genom getestet haben, nach bestimmten Varianten zu suchen, die mit Merkmalen oder medizinischen Bedingungen in Verbindung stehen.
Um es für genetische Genealogen weniger umständlich zu machen, haben DNS-Test-Unternehmen SNPs mit kürzeren Handhabungs-Namen umbenannt. Zum Beispiel wurde die Mutation rs34276300, die den keltischen Zweig der Haplogruppe R1b definiert, durch Family Tree DNA in P312 umbenannt. Die Rivalität zwischen den Testfirmen führte dazu, dass sie ihre eigene Nomenklatur benutzten, so dass P312 von EthnoAncestry S116 genannt wurde. Einige Haplogruppen werden von mehreren SNPs definiert (manchmal Hunderte für alte Haplogruppen, die prähistorische Engpässe hatten). Bei Eupedia sehen Sie normalerweise nur den SNP, der auf phylogenetischen Bäumen verwendet wird, um Verwechslungen zu vermeiden.
=> Lesen Sie mehr Fakten über Genetik
|
Populationsgenetiker haben Zehntausende von Y-DNS-Mutationen klassifiziert, die häufig bei Menschen auf der ganzen Welt gefunden werden, und haben den genealogischen Stammbaum der Menschheit wieder aufgebaut. Im Letzteiszeitlichen Vergletscherungs-Maximum (abgekürzt LGM auf Englisch, vor ca. 19.000 bis 26.000 Jahren), vor allem in Europa, Zentralasien und Nordasien, die zum Teil von riesigen Eiskappen bedeckt waren, gab es für die Menschen einen ausgeprägten genetischen Flaschenhals. Viele Linien starben während dieser Zeit aus. Als die Population wieder zu wachsen begann, trugen Männer, die vom gleichen Stamm stammten, die gleiche lange Reihe von Mutationen auf ihrem Y-Chromosom, die ihr letzter gemeinsamer väterlicher Vorfahre seit mehreren Jahrtausenden angesammelt hatte, bevor der Bevölkerungsengpass auftrat. Genetiker wählten diese Knoten von manchmal über 100 akkumulierten gemeinsamen SNPs, um die wichtigsten prähistorischen Stämme der Welt zu definieren, die sie Haplogruppen nannten. Mit anderen Worten, Menschen, die eine Reihe identischer einzigartiger Mutationen teilen, gehören zu derselben Haplogruppe und stammen von demselben patrilinearen Vorfahren ab. Es ist möglich, zu bestimmen, wann dieser Vorfahr gelebt hat, basierend auf der Anzahl von neuen Mutationen, die seitdem bei gegenwärtigen Individuen aufgetreten sind.
Ursprünglich teilten Populationsgenetiker die Menschheit in 20 Haplogruppen auf, die jeweils in chronologischer Abfolge nach einem Buchstaben von A bis T benannt waren. Haplogruppe A ist die Quelle der Menschheit in Afrika. Für jede neue Einteilung einer Linie wurde nach der Haplogruppe eine Nummer vergeben. Zum Beispiel sind R1 und R2 zwei Zweige der Haplogruppe R. Zahlen und Buchstaben wechseln sich dann für die aufeinanderfolgenden Unterteilungen ab. Zum Beispiel R1a und R1b, dann R1a1, R1a2, R1b1 und R1b2. Einige Zweige ließen fast keine Nachkommen zurück (z. B. R1a2), während andere blühten (z. B. R1a1). Die folgende Tabelle zeigt, wann sich die wichtigsten Haplogruppen in Europa und im Nahen Osten entwickelten.
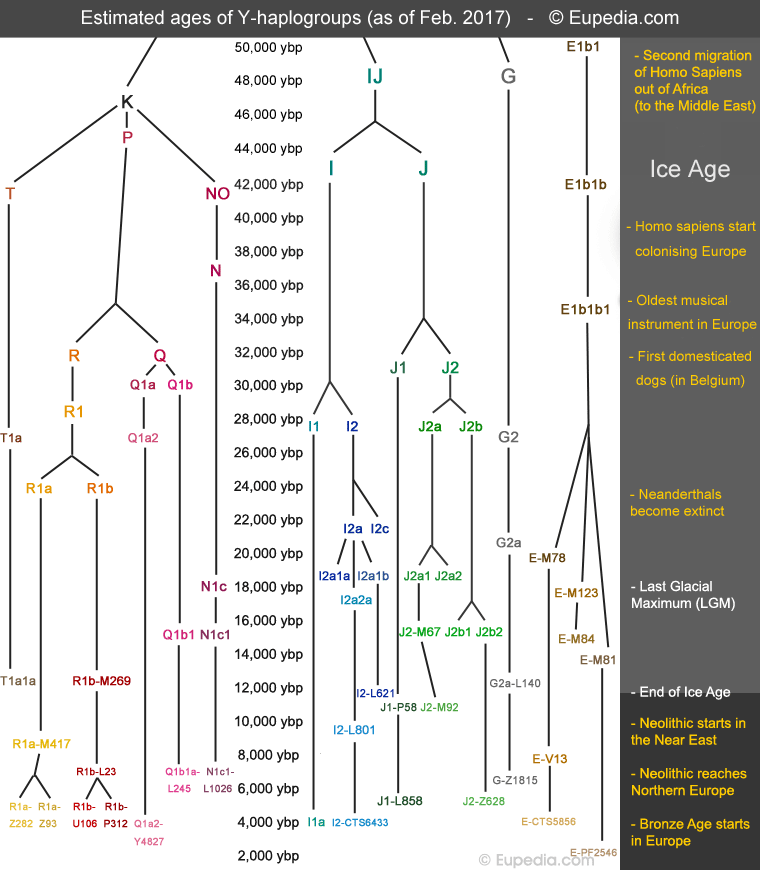
Nach der letzten Eiszeit besiedelten die Menschen die nördliche Hälfte Europas von LGM Refugien in Südeuropa aus. Andere Stämme zogen aus Anatolien und Zentralasien aus nach Europa. Vor etwa 11.000 Jahren wurde im Fruchtbaren Halbmond die Landwirtschaft erfunden. Ein paar Jahrtausende später breiteten sich neolithische Bauern in alle Richtungen aus und vermischten sich mit den mesolithischen Jägern und Sammlern, die zu dieser Zeit in Europa und anderen Regionen lebten.
Vor 5.000 Jahren wurden im Nordkaukasus die ersten Bronzewaffen von Proto-Indogermanen erfunden, die zum ersten Mal in der Geschichte auch Pferde zähmten. Diese Reiter mit Bronzewaffen verließen die Pontische Steppe Südrusslands und eroberten den größten Teil Europas, Zentralasiens und Südasiens.
Während der Bronze- und Eisenzeit entstanden und erweiterten sich die ersten Zivilisationen. Europa sah den Aufstieg und Fall der Kelten, der Griechen, der Römer ... Dann kamen die großen Völkerschaften der alten germanischen, slawischen und zentralasiatischen Stämme, später gefolgt von den Wikingern.
Jede dieser Wanderungen verbreitete neue Gene und neue Y-DNS-Linien, über die Sie hier im Detail lernen können, mit Erklärungen zu den alten Ethnien, die mit jeder Gruppe verbunden sind. Sie können die Y-DNS-Häufigkeiten einfach nach Land und Region vergleichen und die Verteilungskarten für jede Haplogruppe und ihre Hauptuntergruppen visualisieren.
Was ist mit der Mutterlinie?
Dasselbe kann auf der mütterlichen Seite mit mitochondrialer DNS (mtDNA) geschehen. Mitochondrien sind Organellen, die Zellen im Körper Energie liefern. Sie haben ihre eigene DNS, die sich vollständig von der Kern-DNS unterscheidet, die die 23 Chromosomenpaare enthält. Diese mitochondriale DNS wird nur durch Mütter weitergegeben, da das Spermatozoon nach der Zeugung seine mtDNA verliert und der Embryo seine mtDNA von der Eizelle der Mutter erbt.
Obwohl mtDNA die erste genetische Methode zur Rückverfolgung von Vorfahren war, ist ihr Umfang begrenzter, da mtDNA eine viel kürzere Sequenz (16.569 Basenpaare) als das Y-Chromosom aufweist und Mutationen viel seltener vorkommen als auf dem Y-Chromosom. Daher ist mtDNA nur für die Rückverfolgung sehr weit entfernter Herkunft nützlich, typischerweise von vor über 4.000 Jahren (mehr dazu).
Da Mitochondrien die Kraftwerke der Zellen sind, können Mutationen in der mtDNA die Art beeinflussen, wie der Körper seine Energie produziert und nutzt. Einige mtDNA-Haplogruppen sind mit einem effizienteren Sauerstoffverbrauch (VO2 max) und einer größeren körperlichen Ausdauer (z. B. Haplogruppe H) in Verbindung gebracht worden, während andere mit schlechterer athletischer Leistung (z. B. J2 und K) verbunden sind. Es wurde berichtet, dass die Haplogruppen U und K in Cybridzellen einen höheren pH-Wert aufweisen, was Schutz gegen Schlaganfälle und neurologische Störungen verleiht und mit einem etwas höheren IQ korreliert. Die C150T-Mutation, die möglicherweise in jeder Haplogruppe gefunden werden kann, wurde mit erhöhter Langlebigkeit und Stressresistenz in Verbindung gebracht. Es gibt viele andere gesundheitserhebliche Umstände, die mit mtDNA-Mutationen in Verbindung gebracht werden (siehe die mtDNA-Haplogruppenseiten wegen weiterer Einzelheiten), und das kann an sich ein interessanterer Grund sein, seine mtDNA-Haplogruppe zu kennen, als zu Zwecken der Kenntnis der Abstammung.
Wie kann ich meine DNS testen?
DNS-Tests sind sehr einfach. Sie müssen lediglich ein Testkit bei einer Testfirma bestellen, ein Wattestäbchen im Mund reiben (oder je nach Firma in einen kleinen Behälter spucken) und per Post zurückschicken. Die Ergebnisse dauern normalerweise zwischen 6 und 12 Wochen, nachdem das Labor Ihre Probe erhalten hat.
Welchen DNS-Test sollte ich wählen?
Bitte lesen Sie den Hauptartikel:
Autosomale Rechner & GedMatch
Tests uralter DNS sind zu einem unglaublichen Werkzeug geworden, um die Geheimnisse der Urgeschichte und der Völkerwanderungen aufzuklären. Die Genome von Hunderten von Personen aus der Altsteinzeit (einschließlich Cro-Magnons und Neandertalern) bis zum Mittelalter (Wikinger, Magyaren) wurden getestet, und viele dieser Genome wurden öffentlich zugänglich gemacht. Jeder, der seine autosomale DNS getestet hat, kann sie mit jeder dieser alten Proben vergleichen, seien es neolithische europäische Bauern, Proto-Indoeuropäer aus Russland, Eisenzeitkelten, römische Briten oder Angelsachsen, um nur einige zu nennen. Durch das Hochladen der Rohdaten in GEDMatch kann jeder sehen, welche uralte DNS (oder die heutiger Bevölkerungen) ihm genetisch am nächsten steht.
Eine Reihe von autosomalen Rechnern wurde entwickelt, wie das Dodecad Ancestry Project (von Dienekes Pontikos), Eurogenes (von David Wesolowski), Harappa Ancestry Project (von Zack Ajmal), Fennoscandia Biographic Project (von Anders Pålsen), and Magnus Ducatus Lituaniae Project (MDLP) (von Vadim Verenich und Leon Kull), um sein Genom mit Modellen historischer oder regionaler Populationen zu vergleichen. Einige von ihnen verwenden Datensätze antiker DNS als Referenzpopulationen, so dass man beispielsweise abschätzen kann, wie viel Prozent seiner DNS von mesolithischen europäischen Jägern/Sammlern im Vergleich zu jungsteinzeitlichen Bauern und zu Steppen-Indogermanen vererbt wurde. Andere Rechner versuchen, den Prozentsatz der Herkunft zu bestimmen, der mit Y-DNS-Haplogruppen verknüpft ist (z.B. entspricht die moderne R1a-Verteilung in Europa der osteuropäischen Beimischung in Dodecad K12, während R1b westeuropäisch ist). Verbreitungs-Karten sind auf Eupedia für mehrere Komponenten von Dodecad- und Eurogenes-Rechnern verfügbar, erstellt unter Verwendung von Daten von Tausenden von Teilnehmern und akademischen Proben für ethnische Minderheiten (z. B. im Kaukasus).
Sie müssen Ihre DNS-Daten nicht hochladen oder senden, um Ihre Herkunftsschätzung zu erhalten. Alles, was Sie tun müssen, ist das Herunterladen von Do-It-Yourself Dodecad v 2.1 und der Rechner, die Sie ausprobieren möchten, und das Befolgen der Anweisungen. Beachten Sie, dass nicht alle Rechner zum Download verfügbar sind und einige nur über GEDMatch verfügbar sind. Der Do-it-Yourself Dodecad wurde ursprünglich 2011 für 23andMe, Geno 2.0 und Family Finder entwickelt, da LivingDNA und Chromo 2.0 zu dieser Zeit noch nicht existierten. Die Ergebnisse sind mit jedem Test gleichwertig.
Wenn Sie sich nicht für autosomale Berichte von Testfirmen interessieren und nur an autosomalen Rechnern interessiert sind oder Ihr Genom auf GEDMatch vergleichen möchten, ist jeder autosomale Test geeignet. Wenn Sie nicht an Ihren mtDNA-Ergebnissen interessiert sind (die für Vorfahren jünger als die Bronzezeit von begrenztem Wert sind) und bereits Ihre Y-DNS getestet haben oder die vollständige Y-DNS-Sequenz (siehe unten) testen wollen oder, wenn Sie weiblich sind und Y-DNS nicht testen können, können Sie getrost mit dem billigsten Autosomal-Test (MyHeritage oder Family Finder) fortfahren.
Was sind Nachnamenprojekte?
Family Tree DNA hat Tausende von DNS-Familienprojekten, in denen Menschen ihre Y-chromosomale DNS mit anderen Mitgliedern gleichen oder ähnlichen Namens vergleichen können und versuchen, zu bestimmen, welche Mitglieder verwandt sind und wie viele Generationen seit ihrem letzten gemeinsamen Vorfahren verstrichen sind. Um an einem solchen Projekt teilzunehmen, müssen Sie einen speziellen STR-test (Short Tandem Repeats)durchführen, der sich vom SNP-Test von 23andMe, LivingDNA und Geno 2.0 unterscheidet. Familiennamen basieren auf dem Y-Chromosom, da Familiennamen jeweils vom Vater vererbt werden und daher der Y-chromosomalen Abstammung folgen. Nur Männer können diesen Test machen.
Bis vor kurzem bestand der Vorteil darin, dass diese STR-Tests präziser waren, um kürzlich geteilte Herkunft zu schätzen, als grundlegende 'Backbone'-SNP-Tests. Es ist folglich die bevorzugte Methode für genetische Genealogie geworden, besonders für Leute, die ihren Familienstammbaum mit entfernten Verwandten bestätigen oder gewöhnliche Abstammung zwischen Personen feststellen wollen, die den gleichen Nachnamen teilen, aber keinen papierernen Nachweis einer Verbindung haben. Die Entwicklung tiefgehender SNP-Tests mit über zehntausend Markern, ganz zu schweigen von vollständigen Y-Chromosomen-Tests wie Y Elite 2.1, die Millionen von ihnen abdecken, hat jedoch STR-Tests etwas veraltet (und vergleichsweise überteuert) gemacht. Dennoch sind sie immer noch weit verbreitet, da sie die erste Art von Tests in der genetischen Genealogie waren und Nachnamen-Projekte, die auf der Website von Family Tree DNA begonnen wurden, haben Zehntausende von Teilnehmern in den letzten zehn Jahren erworben.
Ihr Test wird Sie nur über Ihre agnatische (patrilineare) Linie informieren, aber nichts hindert Sie daran, andere männliche Familienmitglieder, die einen anderen Nachnamen haben, zu fragen, ob sie einen Test machen wollen. Um die agnatische Linie Ihrer Mutter zu kennen, sollten Sie entweder Ihren Vater (wenn noch am Leben), einen ihrer Brüder oder einen ihrer Oheime väterlicherseits (oder den Sohn eines Oheims väterlichereits) testen. Dasselbe kann mit den agnatischen Linien Ihrer Großmutter geschehen, indem Sie einen ihrer Brüder oder männliche Kinder eines Bruders testen. Verwandte können sogar entfernte Verwandte sein, solange sie männlich sind und denselben Nachnamen tragen.
Nachverfolgen
Stellen Sie Ihre Fragen und diskutieren Sie über Haplogruppen auf dem Forum