Répartition géographique
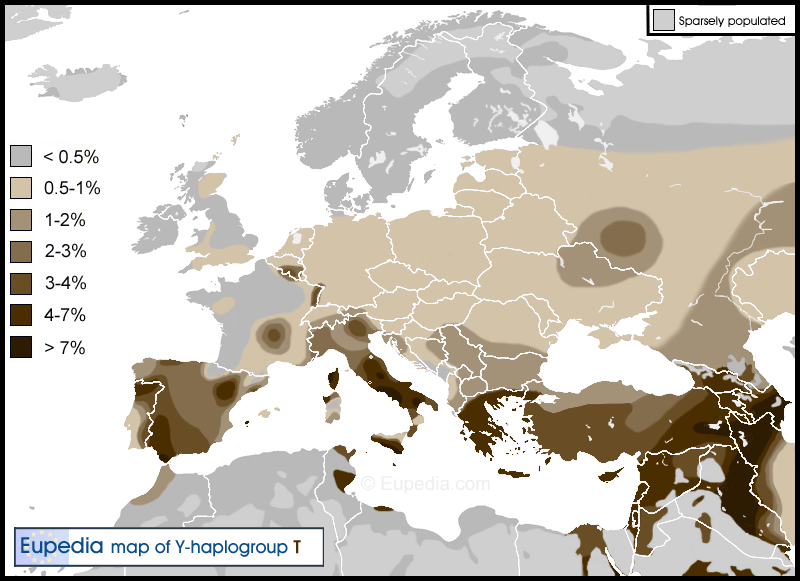
L'haplogroupe T est un lignage assez rare en Europe. Il représente seulement 1 % de la population sur la majeure partie du continent, sauf en Italie, en Grèce et en Macédoine, où il dépasse 4 %, et dans la péninsule ibérique où il atteint 2,5 %, avec des enclaves allant jusqu'à 10 % à Cadix et plus de 15 % à Ibiza. La fréquence maximale de l'haplogroupe T dans le monde est observée en Afrique de l'est (Erythrée, Ethiopie, Somalie, Kenya, Tanzanie) et au Moyen Orient (surtout le Caucase du Sud, le sud de l'Iraq, le sud-ouest de l'Iran, l'Oman et le sude de l'Egypte), où il représente environ 5 à 15 % des lignées paternelles. Outre ces régions et l'Europe, on trouve des poches isolées d'haplogroup T en Asie centrale, en Inde, en Afrique du Sud, au Cameroun et au Zambie. Sa densité la plus élevée se trouve en fait chez le peuple Fulani du Cameroun (18 % de la population).
Répartition géographique de l'haplogroupe T en Europe, en Afrique du nord et au Moyen-Orient
Phylogénénie de T1a
Si vous êtes nouveau en généalogie génétique, veuillez vérifier notre Introduction à la phylogénie pour comprendre comment lire un arbre phylogénétique.


Origines & Histoire
L'haplogroupe T apparu il y a entre 15.000 et 22.000 ans, ce qui en fait un haplogroupe relativement jeune. T est un descendant de l'haplogroupe K, qui est lui-même l'ancêtre de la plupart des haplogroupes eurasiatiques (L, N, O, P, Q, R et T), et dont l'origine probable est au Moyen Orient ou en Asie centrale.
Bien que l'haplogroupe T soit plus fréquent aujourd'hui dans la Corne de l'Afrique que partout ailleurs, il y a de fortes chances que T se soit propagé à partir du Croissant Fertile avec l'essor de l'agriculture. En effet, les sous-clades les plus anciennes et la plus grande diversité de T se trouve au Moyen-Orient, en particulier autour du Croissant Fertile. href="http://www.nature.com/nature/journal/v536/n7617/full/nature19310.html" Lazaridis et coll. (2016) ont identifié un membre de l'haplogroupe T parmi les vestiges du Néolithique précéramique B en Jordanie. Un échantillon T1a a également été trouvé dans le Néolithique culture rubanée (LBK) en Allemagne par href="http://biorxiv.org/content/early/2015/10/10/016477" Mathieson et coll. (2015) . href="https://era.library.ualberta.ca/files/wm117r51m" Nour Mousa a signalé la présence de l'haplogroupe K ( xN1c1-TAT, O-M175, P-M45) parmi les premiers habitants néolithiques de la région du lac Baïkal en Sibérie méridionale. Cet échantillon n'a pas été testé pour les mutations de l'haplogroupe T, mais il s'agit très probablement d'un T1a, compte tenu de sa présence dans la région aujourd'hui et de l'absence d'autre alternative plausible (en dehors de K* et N*).
Colonisation néolithique de la péninsule arabique et de l’Afrique de l’Est
L'incidence plus élevée de T1 en Afrique de l'Est aurait été causé par un effet fondateur parmi les agriculteurs ou d'éleveurs pastoraux néolithiques du Moyen Orient. Une théorie veut que l'haplogroupe T se soit propagé aux côtés de J1 en tant que éleveurs-chasseurs à l'époque néolithique précéramique, partant des monts Zagros entre 9.000 et 10.000 ans avant notre ère, pour atteindre l'Egypte et le sud de la péninsule arabique autour de 7.000 ans avant notre ère, puis de là se seraient étendus à la corne de l'Afrique, et plus tard à Madagascar. Toutefois, compte tenu que J1 a une présence particulièrement forte au Yémen et au Soudan, tandis que T1 est plus fréquent dans le sud de l'Égypte, en Érythrée et en Somalie, les deux lignées ne se sont pas nécessairement propagées ensemble. Elles pourraient plutôt avoir constituées différentes tribus d'éleveurs nomades qui auraient colonisé la région de la mer rouge durant le Néolithique, une période s'étendant sur plusieurs millénaires. Néanmoins, les deux se retrouvent dans toute la péninsule arabique, dans tout le nord-est de l'Afrique, de l'Égypte à la Somalie, ainqi qu'à Madagascar. Cela contraste avec les autres haplogroupes du Proche-Orient tels que G2a et J2, qui se font remarqués par leur absence en Afrique de l'est et leur rareté dans la péninsule arabique.
Colonisation néolithique du sud de l'Europe
Les tests d'ADN préhistorique ont révélé que la majorité des premiers agriculteurs néolithiques qui ont colonisé l’Europe appartenaient à Y-haplogroupe G2a, avec une minorité d’autres haplogroupes tels que C1a2, E1b1b, H2, I2, J2 et T1a. En fait, à présent l'haplogroupe T n'a été identifié que dans les restes de la culture de la céramique linéaire (LBK) en Allemagne. Néanmoins, moins de 100 échantillons néolithiques européens d'Y-ADN ont été testés à ce jour, et il est encore trop tôt pour dire si les lignages G2a dominaient dans toutes les cultures européennes néolithiques ou seulement en Europe centrale et du sud-est.
Nos jours haplogroupe T1a est beaucoup plus courant en Europe méditerranéenne qu’ailleurs sur le continent. Ceci suggère que la plus grande partie des agriculteurs T1a pourrait avoir suivi un itinéraire différent du Proche-Orient, soit en bateau le long des côtes, et/ou par l’intermédiaire de l’Afrique du Nord. Dans la première hypothèse, les agriculteurs T1a auraient été associés à la culture de la céramique cardiale (5000-1500 AEC). Toutefois, aucun des échantillons de cette culture ou d'autres cultures néolithiques plus tardives dans la péninsule ibérique ou en Italie n'ont révélé la présence de T1a (une fois de plus G2a était la lignée dominante). L'itinéraire de colonisation via l'Afrique du Nord est donc plus plausible et expliquerait pourquoi les sous-clades de T1a trouvées en Europe, notamment T1a1a1a1 (CTS2214), T1a1a1b (Y6671), T1a1a2 (Y16897), T1a2a1 (Y6055) et T1a2b1 (P322), sont surtout présentes dans le sud du Moyen Orient (sud du Levant et péninsule arabique) et dans le sud de l'Europe. Il existe peu de données pour l’Afrique du Nord, mais certaines de ces sous-clades ont été confirmés en Egypte (Y6671 > Y12643), en Libye (Y6671 > Y22559) et au Maroc (Y6671 > Y12643).
En outre, au sein de l’Europe, la fréquence de T1a reflète celle de l'haplogroupe J1, comme c'est également le cas dans la péninsule arabique. En Europe, ces deux haplogroupes sont plus fréquents dans les zones montagneuses de la partie sud des Balkans, les parties centrales et sud de l'Apennin en Italie, la Sicile, le Massif Central en France (Auvergne) et le sud-ouest de la péninsule ibérique. Toutes ces régions auraient été mieux adaptées à l'élevage des chèvres et des moutons que pour la culture des céréales dans la période néolithique. Les régions montagneuses permettent aux éleveurs la pratique de la transhumance, c'est-à-dire le mouvement saisonnier des éleveurs et de leur bétail entre des pâturages d’été et d’hiver bien déterminés.
Colonisation néolithique du nord-est de l’Europe et de la Sibérie
T1a2 (L131) a été trouvé aussi loin que la région Volga-Oural russe et Xinjiang dans le nord-ouest de la Chine. Cette branche a probablement pénétré dans la Steppe pontique pendant le Néolithique (peut-être aux côtés de J2b) et s’intégra aux peuples autochtones R1a avant leur expansion vers l’Asie centrale au cours de l’âge du Bronze (=> voir R1a-Z93). La thèse de doctorat de Nour Mousa a signalé la présence de l’haplogroupe K (xN1c1-TAT, O-M175, P-M45) parmi les premiers habitants néolithiques de la région du lac Baïkal en Sibérie méridionale. Ces échantillons n’ont pas été testé pour les mutations définissant l'haplogroupe T, mais il s'agit très probablement de T1a compte tenu de sa présence dans la région aujourd'hui et l’absence d’autre alternative plausible (outre K* et N).
L'haplogroupe T a été trouvé à une fréquence relativement élevée parmi les Tatars (5 %) et les Maris (2 %) de la région Volga-Oural, ainsi que dans le nord-ouest de la Russie (3 %), ce qui suggère qu'il pourrait avoir été un des principales lignages qui a apporté le néolithique aux populations de langue ouralienne. Les tests ADN autosomique ont également identifié des pourcentages anormalement élevés de sang sud-ouest asiatique parmi les Finlandais (1 à 2,5 %) et les Lituaniens (1,5 %), qui n'ont autrement pas d'ADN moyen-oriental ou caucasien et ne possèdent presque pas d'ADN-Y du Moyen Orient. Cet ADN "sud-ouest asiatique" pourrait être la trace des lignées T absorbées pendant le néolithique.
Expansion kouro-araxienne durant l’âge du bronze
Les branches P77 et CTS6507 ont connu une expansion importante au début de l'âge du Bronze, il y a environ 4.500 ans. La phylogénie suggère que cette expansion a eu lieu à partir de la région du Caucase du Sud, y compris le haut-plateau arménien, et s'est dispersée dans diverses directions autour du Moyen Orient et de l'Europe. La branche européenne semble s'être propagée via la Méditerranée vers la Grèce, l'Italie (y compris la Sicile et la Sardaigne) et la péninsule ibérique. Historiquement, la culture kouro-araxienne correspond le mieux à cette expansion. Alors que les Proto-Indo-Européens (haplogroupes R1a et R1b) envahissaient le centre et le nord de l'Europe ainsi que l'Asie centrale à partir de la Steppe pontique, le peuple kouro-araxien, de l'autre côté du Caucase, a également développé une culture contemporaine de l'âge du Bronze qui s'est étendu dans tout le Moyen Orient et peut-être aussi loin que le Pakistan et l'Inde. Les Minoens, la civilisation européenne la plus ancienne (par opposition aux cultures archéologiques), pourrait descendre de cette expansion kouro-araxienne. Les hommes kouro-araxiens auraient appartenu principalement à l'haplogroupe J2a1, mais aussi dans une moindre mesure à G2a-L293, G2a-M406, J1-Z1828, L1b, T1a-P77 et R1b1-L278.
Civilisations anciennes associées à l'haplogroupe T
Lors de l'âge du Cuivre et de l'âge du Bronze, l'haplogroupe T aurrait été un lignage important (bien que probablement pas dominant) parmi des peuples antiques tels que les Sumériens, les Babyloniens et les Assyriens.
Les fréquences d'haplogroupe T plus élevées que la moyenne dans des endroits comme Chypre, la Sicile, la Tunisie, Ibiza, l'Andalousie et la pointe nord du Maroc suggèrent que l'haplogroupe T pourrait s'être disséminé via la colonisation des Phéniciens (1200-800 avant notre ère), et que l'ancienne Phénicie aurait apparemment eu une incidence plus élevée de T que le Liban actuel (5 %).
Les lignées maternelles (ADNmt) correspondant à l'haplogroupe T
Une façon de déterminer les lignées maternelles originales qui auraient accompagné les agriculteurs du début du Néolithique appartenant à Y-haplogroupe T est de comparer les haplogroupes ADNmt trouvés dans les régions avec des fréquences élevées d'haplogroupe T aujourd'hui. Il est particulièrement utile de regarder des régions très éloignées et des groupes ethniques qui ne partagent pas d'autres lignées paternelles que T. Heureusement Les tribus néolithiques T se sont dispersées jusqu'à très loin dans toutes les directions à partir du Proche-Orient, colonisant Europe, Afrique de l'Est, diverses régions d'Inde et même la Sibérie. En analysant les haplogroupes ADNmt des pays d'Afrique orientale avec des fréquences élevées de T, tels que l'Ethiopie, la Somalie ou de parties de Madagascar, et en les comparant avec des échantillons anciens des agriculteurs néolithiques, les meilleures correspondances sont les haplogroupes mitonchondriaux HV, N1a et U3.
Individus T célèbres

Le test de l'ADN présumé du 3e président américain, Thomas Jefferson, a conclu qu'il appartenait à l'haplogroupe T.
Suivi
Posez vos questions et discuter des haplogroupes sur le Forum